카테고리가 대학원생활입니다. 제가 생각하는 KOGES에 대해서 이야기하는것이니 다른 의견이 있으시면 댓글 달아주세요!
KOGES란 질병관리청에서 데이터를 생성하고 있는 한국인 유전체 역학 조사사업으로 국내 최대 규모의 코호트 연구라고 이야기들 합니다.
KOGES에서는 Array데이터를 이용을 하여 대규모 유전체 데이터를 생산하고 있는데, 우리나라에서 개발한 K-chip을 사용합니다.
www.cdc.go.kr/contents.es?mid=a40504030900
질병관리청 국립보건연구원
질병관리청 국립보건연구원
www.cdc.go.kr
위 사이트에서는 KOGES에서 몇명의 데이터가 있는지 대략적으로 알수가 있습니다.
생각보다 많은 데이터를 갖고 있지만, 저도 혹은 주변에서 KOGES 데이터를 사용도 해보았고, K-chip도 사용을 해보았지만 조금 회의적인 생각이 있습니다.
소규모로 K-chip을 약 100 Sample미만으로 해본적이 있는데, QC도 회사에서 맡겨서 왔었습니다. 그리고 분석후에 질병 후보 변이 몇개를 찾았었습니다. (자체 QC분석과 함께 같이 분석하였습니다.)
그리고 그 변이에 대해서 확인하기 위해서 Sanger sequencing을 맡겼는데, 나와야할 변이가 나오지 않는 것입니다. Sanger sequencing으로 확인하려던 샘플의 50%이상에서 나와야할 변이가 나오지 않는 것을 확인하였고, 거기서 처음으로 의심이 들었습니다.
저는 Microarray에 대해서 자세히는 모르지만, Genotype을 결정을 하는데 중간 Clustering을 통하여 해당변이의 genotype을 구하는 것으로 알고 있습니다.
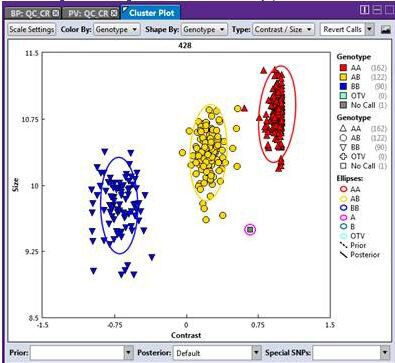
샘플이 많으면 많을수록 정확한 Clustering이 된다고 array 회사에서 이야기를 하는데, KOGES의 경우 수만명의 데이터가 있을 것이고, 그 중에서 특정변이에서 Cluster가 경계에 있는 샘플들의 경우는 어떻게 처리가 될까 라는 생각이 들었습니다. 그 경계에 있는 샘플들 정말 변이가 정확할까? 라는 생각이 들었습니다. (여기서 Array회사에서 제시하는 Accuracy를 이야기하면 딱히 할말이 없습니다. 저도 그것을 알고 있지만 실제 Array분석을 해보니 들었던 생각입니다.)
아까 제가 분석했던 이야기로 다시 돌아와서 해당 변이에 대해서 Array를 맡겼던 회사에 연락을 취했습니다. 그쪽 회사에서는 샘플이 적어서 그렇다 그리고 Genotyping cluster plot을 보면 Cluster가 Group 형성이 잘 안된것같다 라고 이야기했습니다. (실제로 애매한 cluter를 확인하였습니다.)
그렇다면 또 드는 생각은 KOGES 데이터는 변이 cluster를 하나하나 확인 했을까 라는 생각입니다. 수만명의 데이터를 찍었다 하더라도, Cluster가 잘 되지않는 변이가 있을 것 같은데 이것을 확인을 하지 않았다면 분양받는 연구자는 약간의 리스크를 갖고 분석할수 있을 것 같다는 생각이 들었습니다. (믿고 얻어가야할 공공데이터가 말이죠, 여기서 UK biobank도 Array다. 생각 할 수가 있는데 제 생각은 만들거면 따라서 하기보다는 더 나은 데이터를 생산하는게 더 낫지 않았을까 라는 생각입니다. )
더군다나, KOGES의 경우 분양할때 Imputation 된 것을 분양합니다. 여기서 제가 들었던 의심은 과연 Imputation이 정확할까? 라는 생각입니다. 우리나라 사람들의 AF로 만든 Imputation 데이터 없는 것으로 알고 있습니다.(공개된것중) 대부분 Imputation의 경우 주로 1000Genome에 대한 아시아인의 정보를 이용하는 것으로 알고 있는데, 과연 1000Genome의 아시아인 데이터가 한국인 데이터를 예측하는데 정확할까라는 생각도 드네요. 기억으론 질병관리청에서 만든 한국인 397명에 대한 데이터를 Imputation 정보로 추가했다는 것으로 알고 있는데, 한국인 397에 대한 정확도도 확인이 안된것 같고 뭔가 깨림칙한 느낌이 들었습니다.
약간 정확하지 않는 방법(Microarray Genotyping)으로 나온 결과를 약간 정확하지 않는 방법(Imputation 1000G? or 한국인 397명?)으로 또 다시 예측한 데이터를 KOGES라고 저는 생각합니다. (지금까지 들었던 의심이 말이죠)
KOGES 데이터 10만명 할거 5만명으로 줄이고 Whole Exon sequencing NGS를 했더라면 더 좋았을 것 같단 생각이 듭니다. 아니면 한국인 데이터를 위한 Imputation tool 공개하여 여러 사람들이 사용하도록 했으면 합니다. 데이터 공개되는 것이 문제라면 미시간 imputation server처럼 사용하도록 말이죠.
KOGES에 나쁜 감정은 없습니다. KOGES는 우리나라에서 가장 큰 빅데이터베이스중 하나이고, 데이터를 얻기 위해서는 조금 귀찮지만 잘만 이용한다면 정말 많은 논문들이 나올 수 있을 것 같습니다. 단지 제 모자란 지식 때문인지는 모르겠지만, 제 기준으로 사용하기가 조금 깨림칙하다는 생각이 듭니다. (그렇다고 사용 안할거다라는게 아닙니다. ㅎㅎ)혹시 제 의심에 대해서 저를 가르쳐주실 연구자분! 댓글달아주세요! 관련 논문이라도 던져주신다면 읽겠습니다.
'대학원 생활 전과 후' 카테고리의 다른 글
| 한국생명정보학회 워크샵 정리중입니다. (0) | 2021.02.23 |
|---|---|
| 어후 현재 내용만으로는 블로그 운영이 안될것같네요.. (0) | 2021.02.10 |
| 대학원 생활을 하면서 생각한 것. (0) | 2020.11.18 |
| Shiny 시작! (0) | 2020.09.11 |
| 연구를 하면서 느낀 단점들 (0) | 2020.08.15 |
댓글