오늘은 Genetic risk score (GRS)에 대한 이야기와 실습을 해보고자 합니다.
GWAS 기본 분석은 정말 간단하게 이루어지는 연구입니다.
단지 Case, Control간에 chi-test를 이용하여 변이들의 p-value를 확인하면 되는 부분이죠.
이렇게 반복 결과만 이용해서 논문을 내다 보니 출간 논문에는 특별한 것이 없어지고, 샘플수에 더 초점이 맞춰졌던 것 같습니다.
(이것 이외에도 샘플수에 초점이 맞춰진 이유는 다양합니다. 통계적인 파워 때문일 수도 있고, 특정 소수에게만 적용되는 변이일수도 있기 때문에 샘플수는 GWAS분석에 있어서 중요합니다.)
그러다 사람들이 변이의 p-value를 확인하는 것 이외에 생각을 한 것이 실제 찾은 변이들의 효과를 평가하는 것이었습니다.
"이러한 SNP을 이용할 경우 Control 대비 차이가 난다." 와 같은 찾은 변이를 이용하여 점수를 매김으로서 찾은 변이가 특별하다 라고 이야기 할수가 있을 것 입니다.
이렇게 찾은 변이들을 이용하여 점수를 매기는 방법중 하나는 GRS입니다.
GRS는 여러가지가 연구가 되어있고, 앞으로도 다양하게 나올수 있을 것으로 생각됩니다. (연구주제로서도 좋다고 생각합니다.)
GRS의 경우 아래와 같은 방법이 있습니다.
1. 일반적인 방법

2. weighted 적용 방법
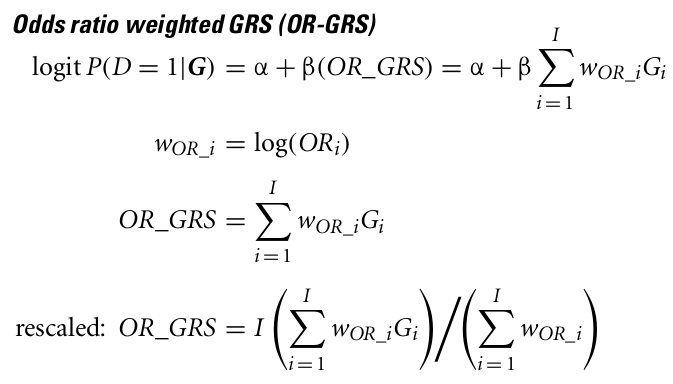
3. maf+or 이용하는 방법

4. etc
위와 같은 방법 중에 우리는 일반 GRS와 weighted GRS에 대해서 실습을 해보도록 하겠습니다.
실습데이터는 다른 포스팅에서 했었던 이명관련 데이터를 사용하도록 하겠습니다.
plink에는 score를 구하는 --score가 있습니다.
--score에 들어가는 파일안에는 SNP, Alt, score가 들어갑니다.
여기서 말하는 score는 weight라고 생각하시면 될 것 같습니다.
실습에 들어가기 전에 score에 들어갈 input file을 만들기 위해서 실습데이터를 살짝 분석해보도록 하겠습니다.
plink --bfile ARHI_IMPUTED_CLEANED --logistic --out ARHI_IMPUTED_CLEANED_logi
위와 같이 logistic regression을 통해서 결과를 보게되면

다음과 같은 결과를 얻을 수 있습니다. 여기에서 p-value cutoff를 진행하고 우리가 필요한 것은 SNP, A1, OR입니다.
연습데이터의 경우 결과에 5e-08에 해당하는 변이가 나오지 않았기 때문에 cutoff를 낮춰서 분석하도록 하겠습니다. (cutoff : 5e-06)
낮춘 cutoff 를 이용하여 결과를 보면 21개의 변이를 확인할 수가 있고, 여기에서 SNP, A1,OR를 뽑아내도록 하겠습니다.
tail cutoff_5e06.txt -n+2 |awk '{print $2,$4,$7}' > score_snplistcutoff_5e06.txt - > 결과에서 cutoff한 파일입니다.
tail을 이용해서 header를 제외하고 awk를 이용하여 2번째, 4번째, 7번째 컬럼을 가져온 것을 보니 다음과 같습니다.

그리고 바로 --score를 이용하여 분석을 해보겠습니다.
plink --bfile ARHI_IMPUTED_CLEANED --score score_snplist --out GRS_Step1
결과 파일은 GRS_Step1.profile에 저장이 됩니다.

결과를 보시면 FID = Family id, IID = Within-Family ID, PHENO = Pheno type (fam 6번째줄), CNT = 찾은 변이 *2, CNT2 = IID안에 변이의 개수, SCORE = GRS(default : mean)입니다.
여기서 결과는 Weighted-GRS이고, 그냥 일반적인 GRS를 구하기 위해서는 OR에다가 1을 넣어주시면 됩니다.
이렇게 해서 구한 점수들을 Case와 Control간 비교를 하면서 자기가 찾은 변이들을 평가할 수가 있습니다.
참고로 plink에서는 missing genotype에 대해서는 imputation을 통해서 예측한 값을 추가로 넣는다고 합니다.
그리고 위에서 default로 구한 값의 경우 평균값입니다.
이를 방지하기 위해서 다음과 같이 분석을 진행해주시면 됩니다.
# Sum GRS
plink --bfile ARHI_IMPUTED_CLEANED --score score_snplist sum --out GRS_Step1_sum
# no mean imputation
plink --bfile ARHI_IMPUTED_CLEANED --score score_snplist no-mean-imputation sum --out GRS_Step1_sum
만약에 두 개다 적용하고 싶을 때는 plink2를 사용하시면 된다고 합니다.
주로 점수를 sum한 것을 사용하는것 같습니다. 그리고 violin plot을 이용해서 차이를 확인하는 것이죠.
그리고 logistic결과 이외에도 quantitative trait에 대한 분석에서도 linear model을 사용하여 분석한뒤 결과에 beta값을 OR 대신으로 넣는 경우도 있었습니다.
여기까지 GRS, weighted-GRS를 구하는 방법에 대해 포스팅 해보았습니다. 감사합니다.
다음에는 PRS에 대해서 tutorial을 포스팅 해보도록 하겠습니다.
'유전체 > GWAS' 카테고리의 다른 글
| [GWAS] 두 vcf 파일을 합칠 때 ref가 다를 경우 어떻게 해야할까? (0) | 2021.06.24 |
|---|---|
| [R,GWAS] 카이검정 (chisq.test), 피셔검정(fisher.test) 쉽게 하기 (0) | 2021.03.09 |
| [GWAS] Imputation 시에 중복된 변이의 경우에는? (0) | 2021.03.03 |
| [GWAS] 결과 분석하는 방법 (assoc function) (17) | 2021.02.01 |
| [GWAS] Genotyping microarray 분석 시 주의해야할 concordance rate (1) | 2020.12.30 |


댓글