저번 heatmap관련 포스팅에서 들어온 질문이 원하는 그룹을 가져오고 싶다 라는 질문이 있었습니다.
간단하게 원하는 그룹을 가져오는 방법에 대해서 설명하도록 하겠습니다.
TCGA - CESC의 데이터를 분석하도록 하겠습니다.
저번과 같이 상위 몇개만을 일단 가져오겠습니다.
CESC<-read.table(gzfile('./CESC/HiSeqV2.gz'),
sep='\t',header=T,stringsAsFactors = F,check.names = F)
library(pheatmap)
CESC1<-CESC
rownames(CESC1)=CESC[,1]
CESC1<-CESC1[1:200,2:ncol(CESC1)]
pheatmap(CESC1)
다음과 같이 heatmap이 그려졌습니다. 여기서

빨간색부분에 대한 자료를 원한다고 가정하고 가져오기 위해서는 다음과 같이 해야합니다.
pheatmap(CESC1,cutree_rows = 4,fontsize_row = 6)

먼저 나누어지는지 확인을 하고, row에 들어간 cluster와 동일한 방식으로 raw data(CESC)를 분석해줍니다.
hclust를 이용할 것이고, hclust의 경우 dist와 같이 사용하셔야 됩니다. hclust다음으로 cutree를 이용할 것 입니다.
hc<-hclust(dist(CESC1))
plot(hc,hang=-1,cex=.5)
rect.hclust(hc,4)
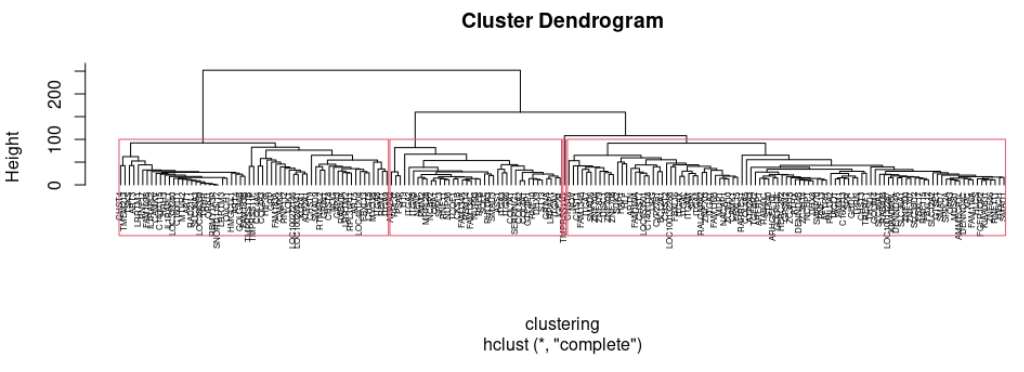
먼저 hclust를 이용해서 그림을 그려보면 heatmap과 유사한 dendrogram을 확인할 수가 있습니다. (잘 보시면 같습니다. 실제로 해보세요.)
그리고 cutree를 하기 전에 hcluster를 어떻게 자를것인지 보여주는 rect.hclust를 이용하면 우리가 pheatmap에서 그렸던 것과 동일한 group으로 나누어지는 것을 확인할 수가 있습니다.
그리고 cutree를 이용하여 그룹을 나누고 개수를 확인해보면 다음과 같이 그룹마다 몇개의 유전자를 포함하는지 알수가 있습니다.
그중에서 우리가 원하는 그룹은 3번째로 큰 그룹이기 때문에 group3을 뽑아주시면 됩니다.
hc_cut<-cutree(hc,4)
table(hc_cut)
#
hc_cut
# output
ZC3H15 TMEM213 C14orf118 ARTN ART1 HRH3 ART3 HERC2P2 ART5
1 2 1 1 2 2 2 1 2
ART4 RNF111 .... 생략
2 1
#output
hc_cut
1 2 3 4
99 61 39 1
> hc_cut[hc_cut==3]
RNF10 RNF11 GTF2IP1 PMM2 ASS1 RBM14 NCBP2 RPL37 DHX9 TCOF1 NUP98 GRINA NUP93 SPPL3
3 3 3 3 3 3 3 3 3 3 3 3 3 3
OPA1 RHEB COL7A1 PSAP ATP2A2 ITGA2 ITGA3 ITGA5 ITGA6 PHLDA3 NENF SQLE MRPL28 H19
3 3 3 3 3 3 3 3 3 3 3 3 3 3
COL4A2 COL4A1 ITGAV FAM134C FAM134A SERPING1 LRPAP1 SMAD3 IGF2R CKS1B ST13
3 3 3 3 3 3 3 3 3 3 3
그리고 정말 내가 원하는 유전자 그룹을 뽑았냐?를 확인하기 위해서는 다음과 같이 bar를 추가해주시면 됩니다.
group1<-data.frame(hc_cut)
group1[group1!=3]=1
group1
pheatmap(CESC1,cutree_rows = 4,fontsize_row = 6,scale = 'none',annotation_row = group1)
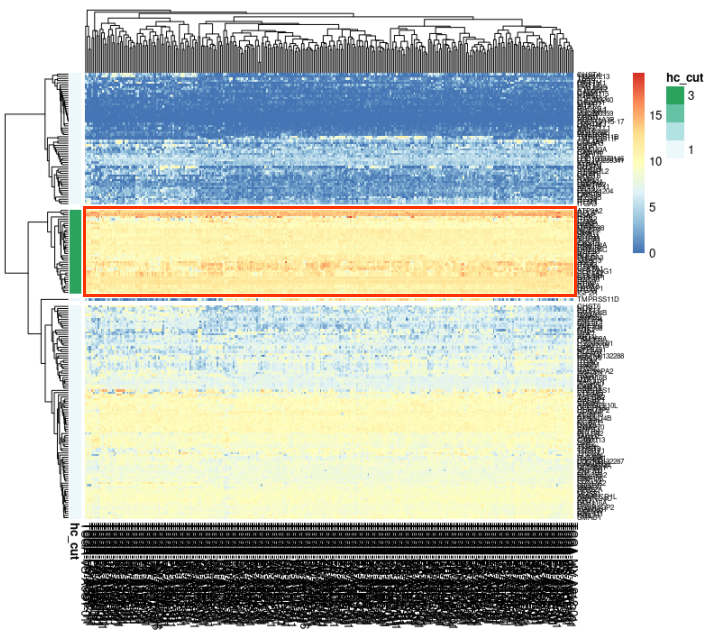
여기까지 heatmap에서 원하는 그룹의 유전자를 가져오는 방법에 대해 포스팅 해보았습니다.
heatmap에서 확인할 수 있는 것은 저 낮은 유전자들은 어떤 GO에 속하는지 확인 하는 방법과 샘플들이 어떤 clustering에서 tumor가 많다 뭐 이런식의 분석이 추가로 가능합니다.
Hclust는 정답이 없는 grouping 방법이라는 것도 생각하셔야 합니다. 그렇기 때문에 cutree의 경우 수학적인 순서로 그룹을 나누어 주고 내가 원하는 그룹이 나올때까지 클러스터를 나누어 줘야합니다.
오늘은 저번 heatmap 포스팅에서 질문하신 내용을 바탕으로 급작스럽게 포스팅 해보았습니다.
뭔가 생각이 정리 하고 글을 쓰는데 너무 갑자기 쓰는 바람에 내용전달이 잘 되었는지 모르겠네요.
모르시는부분 꼭 질문해주시길 바랍니다. (같이 알아가면 좋잖아요!)
'기본적인프로그래밍 > R' 카테고리의 다른 글
| [R] RColorBrewer 이용해서 색감을 확인해보자. (feat. pheatmap) (2) | 2021.12.21 |
|---|---|
| [R] bool로 받기 (na인지 아닌지, 대소문자인지 아닌지 확인) (0) | 2021.10.19 |
| [R] /usr/bin/ld: cannot find -lgfortran error 해결하기 (0) | 2021.03.30 |
| [R] ggplot 산점도 만들 때 순서 정하기. (0) | 2021.03.10 |
| [R] rcdk설치시 오류 해결방법 (0) | 2021.01.29 |



댓글