Expression 연구를 하다가 보면 많이 사용하는 그림이 heatmap입니다.
heatmap의 대표적인 용도는 보여주기식(우리가 발현한 유전자들이 대략 그림처럼 cluster가 되어있다!!)이 강합니다.
뭔가 두 그룹이 다르다는 것을 t.test pvalue이외에도 사람들이 알기 쉽게 하기 위해서는 이러한 heatmap이 필요합니다.
heatmap은 기본적인 heatmap, gplots package에 heatmap.2 기능이 있습니다. 그리고
사람들이 많이 사용하는 ggplot2에서도 만들 수가 있구요.
그러나 오늘은 아주 예쁘게 heatmap을 만들기 위해 주로 사용하는 pheatmap(pretty heatmap)에 대해 실습을 하고자 합니다.
UCSC XENA 데이터를 이용하여 분석을 할 것입니다.
1 . preprocessing 단계
BRCA<-read.table(gzfile('/HiSeqV2.gz'),sep='\t',quote = '',header = T,stringsAsFactors = F,check.names = F)
rownames(BRCA)=BRCA[,1]
BRCA<-BRCA[,2:ncol(BRCA)]
BRCA_con<-BRCA[,grep('-11',colnames(BRCA))]
BRCA_case<-BRCA[,grep('-06',colnames(BRCA))]
BRCA_r<-cbind.data.frame(BRCA_con,BRCA_case)
BRCA_r<-BRCA_r[1:200,]
dim(BRCA_r)
코드를 보면 아시다 시피 정상세포와 전이 샘플을 가지고 왔습니다.
그리고 유전자는 그냥 위에 200개를 가져왔습니다. (데이터가 너무 많으면 그리는게 오래걸리더라구요. )
2. pheatmap 만들어보기
가장 기본적인(default)로 만들어 보겠습니다.
library(pheatmap)
pheatmap(BRCA_r)

뭔가 유전자 사이에 이쁘게 cluster가 된 것을 볼 수가 있네요.
heatmap을 그릴때 row, column사이 hclust를 default로 적용시켜서 보여줍니다.
여기에서 추가적으로 샘플의 정보를 더 넣어 볼까요?
# preprocessing에서 cbind로 붙였기 때문에 rep을 이용하여 쉽게 annotation 파일을 만들수가 있었습니다.
# Sample ID하고 정보하고 매칭을 시켜야합니다.
col_ann<-cbind.data.frame(type=c(rep('Normal',ncol(BRCA_con)),rep('Meta',ncol(BRCA_case))))
rownames(col_ann)<-colnames(BRCA_r)
# annotation_col 추가!
pheatmap(BRCA_r,annotation_col = col_ann)

각 column별로 어떤 샘플들이 cluster가 되었는지 확인이 가능합니다.
여기서 column cluster를 제거 하기 위해서는 다음과 같이 하시면 됩니다.
pheatmap(BRCA_r,annotation_col = col_ann,cluster_cols = F)

그렇게 되면 정상샘플과 전이 샘플은 그대로 두고 유전자 사이 어떻게 cluster가 되는지 확인이 가능합니다.
여기서 더 예쁘게 만들기 위해서는 각 group별로 cluster를 진행후에 matrix를 만들어서 그리면 됩니다.
BRCA_con<-BRCA_r[,grep('-11',colnames(BRCA_r))]
BRCA_case<-BRCA_r[,grep('-06',colnames(BRCA_r))]
hc1<-hclust(dist(t(BRCA_con)))
hc1_c<-cutree(hc1,3)
names(hc1_c)==colnames(BRCA_con)
hc2<-hclust(dist(t(BRCA_case)))
hc2_c<-cutree(hc2,3)
BRCA_r1<-cbind.data.frame(BRCA_con[,names(hc1_c)[hc1_c==3]],BRCA_con[,names(hc1_c)[hc1_c==2]],BRCA_con[,names(hc1_c)[hc1_c==1]],
BRCA_case[,names(hc2_c)[hc2_c==3]],BRCA_case[,names(hc2_c)[hc2_c==2]],BRCA_case[,names(hc2_c)[hc2_c==1]])
colnames(BRCA_r1)[grep('BRCA',colnames(BRCA_r1))]="TCGA-BH-A1ES-06"
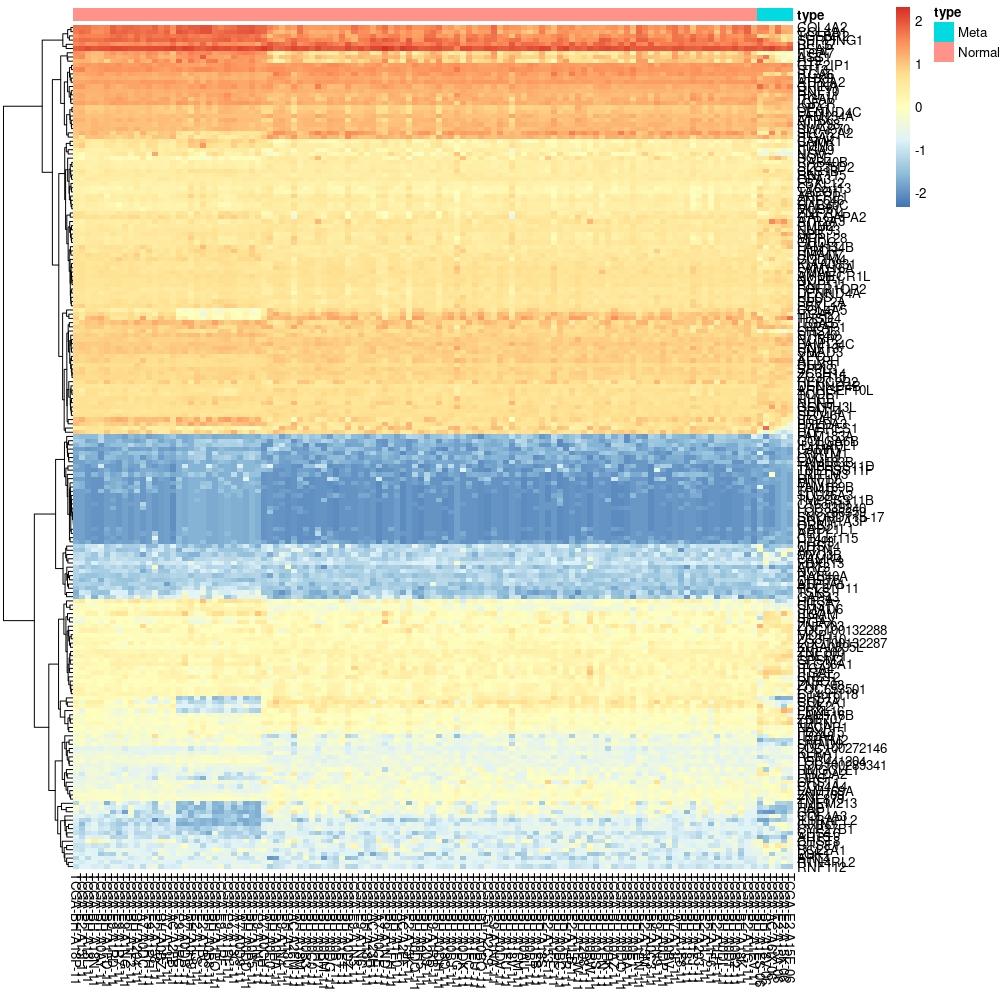
pheatmap(BRCA_r1,annotation_col = col_ann,cluster_cols = F)
뭔가 그럴싸 하죠 ?!
3. 심화 과정
심화과정으로 먼저 normalization을 진행하고
옆에 continuous legend의 범위를 변경해보도록 하겠습니다.
#1. 정규화과정
pheatmap(BRCA_r1,annotation_col = col_ann,cluster_cols = F,scale = 'column')
#2. continuous legend range변경
pheatmap(BRCA_r1,annotation_col = col_ann,cluster_cols = F,scale = 'column',breaks = c(-3,3))
#3. pheatmap확인
help("pheatmap")

정규화를 시킨건 좋았는데, continuous legend를 변경시켜주니 이상한 그림으로 변했습니다.
이럴때는 어떻게 해야할까요?
help(pheatmap)을 쳐보면 default옵션에 color = colorRampPalette(rev(brewer.pal(n = 7, name = "RdYlBu")))(100) 라는 옵션이 보일겁니다.
이 옵션을 그냥 명령어창에다 치면 brewer.pal 에러가 뜨는데, library(RColorBrewer)를 쳐주시면 에러를 해결할수가 있습니다.
그리고 다시 colorRampPalette(rev(brewer.pal(n = 7, name = "RdYlBu")))(100) 를 쳐주시면 100개의 이상한 언어가 나올 것입니다.
이런것으로 보았을 때, 그림이 이상하게 나온 원인은 개수가 안맞는 경우일 것 같습니다. 그래서 조금 맞춰주면 다음과 같습니다.
#1. 혹시 범위로 설정 안해서 그림이 이상하게 나온것 같아서 변경해보았으나, 똑같이 이상하게 나오네요.
c(-3,3) -> c(-3:3)
pheatmap(BRCA_r1,annotation_col = col_ann,cluster_cols = F,scale = 'column',breaks = c(-3:3))
#2. 원하는 구역만 변경시키기
pheatmap(BRCA_r1,annotation_col = col_ann,cluster_cols = F,scale = 'column',breaks = c(-3,-2,-1,0,1,2,3),colorRampPalette(rev(brewer.pal(n = 7, name = "RdYlBu")))(7))
#3. break만 변경
pheatmap(BRCA_r1,annotation_col = col_ann,cluster_cols = F,scale = 'column',breaks = seq(from=-3,to=3,length.out = 100))
1번 그림

2번 그림
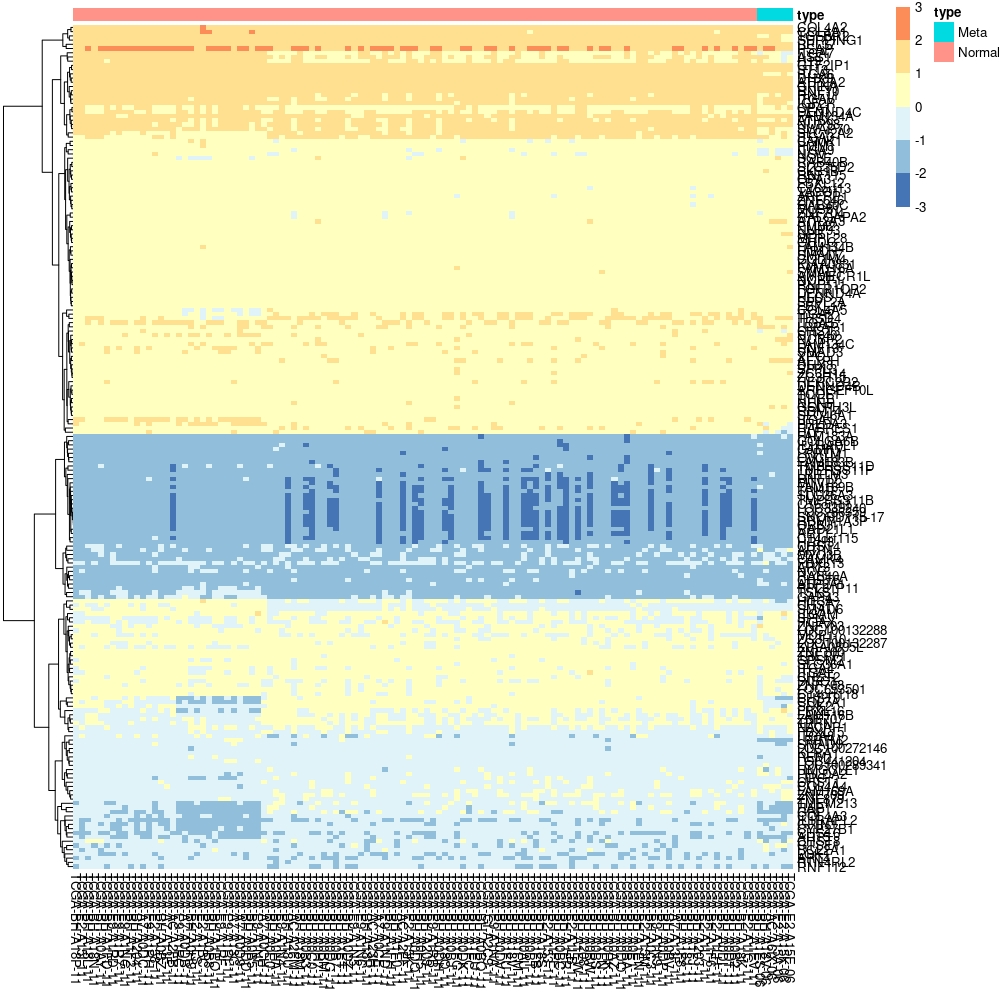
3번그림

다음으로 옆에 hclust를 변경하고, clust에서 그룹을 나누어 보겠습니다.
#'correlation', 'euclidean', 'maximum', 'manhattan', 'canberra', 'binary', 'minkowski'
pheatmap(BRCA_r1,annotation_col = col_ann,scale = 'column',cluster_cols = F,clustering_distance_rows = 'correlation')
# "ward.D", "ward.D2", "single", "complete", "average" (= UPGMA), "mcquitty" (= WPGMA), "median" (= WPGMC) or "centroid" (= UPGMC)
pheatmap(BRCA_r1,annotation_col = col_ann,scale = 'column',cluster_cols = F,clustering_method = 'average')
# cutree
pheatmap(BRCA_r1,annotation_col = col_ann,scale = 'column',cluster_cols = F,clustering_distance_rows = 'maximum',cutree_rows = 5)
hclust를 하기 전에는 distance를 구해줘야합니다. 그 방법은 'correlation', 'euclidean', 'maximum', 'manhattan', 'canberra', 'binary', 'minkowski' 이 있습니다.
여기에서 default는 euclidean입니다. (기본 hclust도 default가 euclidean입니다.)
hclust의 방법도 다음과 같이 여러가지가 있습니다. "ward.D", "ward.D2", "single", "complete", "average" (= UPGMA), "mcquitty" (= WPGMA), "median" (= WPGMC) or "centroid" (= UPGMC)
UPGMA의 경우 계통도에서 많이 사용하는 것으로 알고있고, 뭐 나머지는 잘 모르겠네요!
위와 같이 여러 종류가 있는데 무엇을 써야하냐? 여러가지 돌려보고 cluster가 잘되는 것을 사용하시면 됩니다.
위에서 이야기했듯이 heatmap은 내 자료가 이렇게 잘 cluster가 되었다는 것을 보여주기 위한 그림이기 때문에 괜찮은 cluster 레시피를 확인하고 논문에다 기재하면 되는거죠.
1번 그림

2번 그림

3번그림

여기까지 pheatmap에 대한 포스팅을 완료하도록 하겠습니다. 감사합니다.
필요한 내용 생각나면 업데이트하겠습니다.~!
'실용적인프로그래밍 > R' 카테고리의 다른 글
| [R error] the given url does not appear to be a valid cran repository (0) | 2021.05.13 |
|---|---|
| [R] pheatmap에 오류발생 (NA/NaN/Inf in foreign function call) (5) | 2021.04.19 |
| [R] ggplot barplot, histogram 그리기 (0) | 2021.01.14 |
| [R] ggplot package boxplot 그리기 (0) | 2021.01.13 |
| [R][Shiny] shiny 앱 실행시 발생하는 에러 해결! (0) | 2020.12.18 |


댓글