저번 포스팅은
Plink input format (형식확인)
plink를 설치를 했으면, 설치폴더에 toy라는 파일들이 보입니다. toy.ped, toy.map 이렇게 두 파일의 경우 기본적인 plink input file format입니다. 샘플수가 많을경우 binary파일로 바꿔줘야하는데, 그렇게 바
mopipe.tistory.com
plink input file에 대해서 알아보았고,
이번 포스팅 내용은 input file 만드는 법! 혹은 plink파일에서 vcf를 만드는 방법!!을 알려드리겠습니다!
연습데이터로는
A Pilot Genome-Wide Association Study Identifies Potential Metabolic Pathways Involved in Tinnitus라는 논문에서 (추후 본데이터를 이용하여 GWAS실습을 진행하겠습니다.)
공개해놓은 데이터입니다! 다운받으시면 1.4기가 정도 됩니다!
압축을 푸시고
파일들을 확인해보면 plink binary format으로 되어있습니다! (bed,fam,bim family~!)
plink --bfile ARHI_IMPUTED_CLEANED --out tinnitus --recode vcf을 써주시게되면!
tinnitus.vcf가 생성되게 됩니다!
데이터를 확인해보니 1천명정도되는 샘플데이터이기 때문에 오래걸립니다.
이런 대규모 분석을 위해서는 컴퓨터는 꼭 좋은 것을 사용하시기 바랍니다. 램도 많이 필요하고, 계산이 많이 필요하기 때문에
똥컴들은 느리고 멈출수도있습니다 그리고 컴퓨터의 수명이 더 빨리 닳수있습니다.
어떤 BI분석이든간에 좋은 컴퓨터를 추천드립니다. (최소 I7은 씁시다!! 교수님들에게 징징징!!)

plink format -> vcf를 해보았고, 반대로도 가능합니다. vcf -> plink format
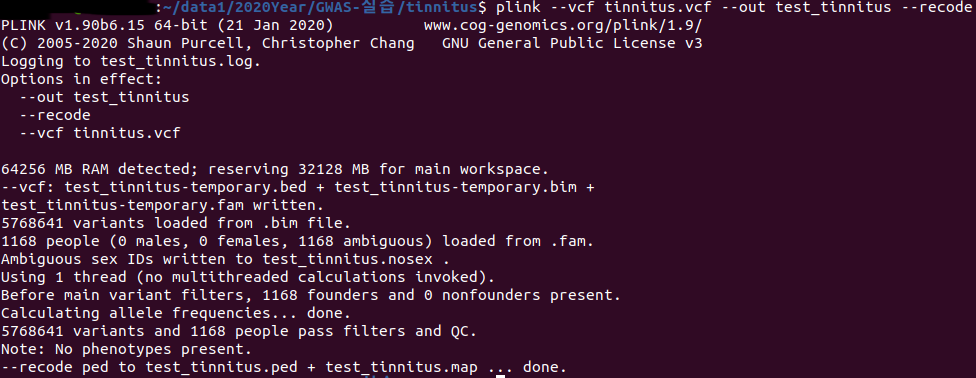

plink --vcf tinnitus.vcf --out test_tinnitus --recode (output ped, map파일)
plink --vcf tinnitus.vcf --out test_tinnitus --make-bed (bim, bed, fam 파일)
이렇게 plink input파일을 vcf로 변환시켜보았고, 반대로 vcf를 plink input으로 바꾸어보았습니다.!
다음 포스팅은 무엇을 할까... 고민이되네요! QC하는 방법은 제가 좀더 공부를 한다음에 포스팅을 해야할것같습니다.
아무튼!! 다음 포스팅도 봐주시길!
참고! 유의점!! vcf에서 Alt에 여러 변이들을 갖는 경우가 있습니다. 이럴 경우 plink format으로 변환하면 missing variant로 처리됩니다. 분석할 때 주의하시기바랍니다!
'유전체 > GWAS' 카테고리의 다른 글
| [GWAS] Plink 유용한 기능 (2) (freq, hardy) (3) | 2020.06.10 |
|---|---|
| [GWAS] plink 유용한 기능 (1) (exclude, extract) (1) | 2020.06.09 |
| [GWAS] plink input format (형식확인) (0) | 2020.06.05 |
| [GWAS] plink 설치 및 설정하기. (0) | 2020.06.04 |
| [GWAS] 시작 (0) | 2020.06.03 |




댓글