어제 포스팅에 이어서 오늘도 Plink 에서 유용한 기능 두번째!!
plink를 이용해서 기본적인 통계기능들을 살펴보도록 하겠습니다.
일단 연습 문제를 만들기위해서!

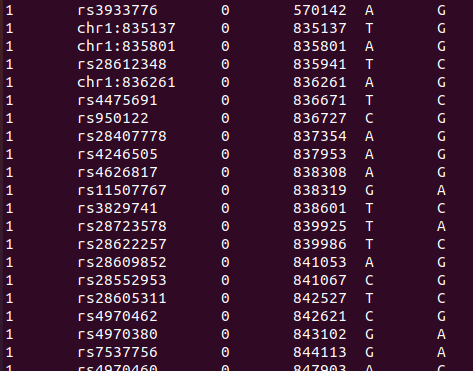
다음과 같이 테스트 샘플을 만듭니다.
Raw data = 이전 포스팅했던 이명관련 open data를 이용하였습니다.
파이썬이나 기타 방법으로 테스트 샘플을 만드시던지 아니면 raw data를 이용하세요! (그런데 오래걸릴 수 있습니다.)
1 . --freq[x]
분석을 진행할때,
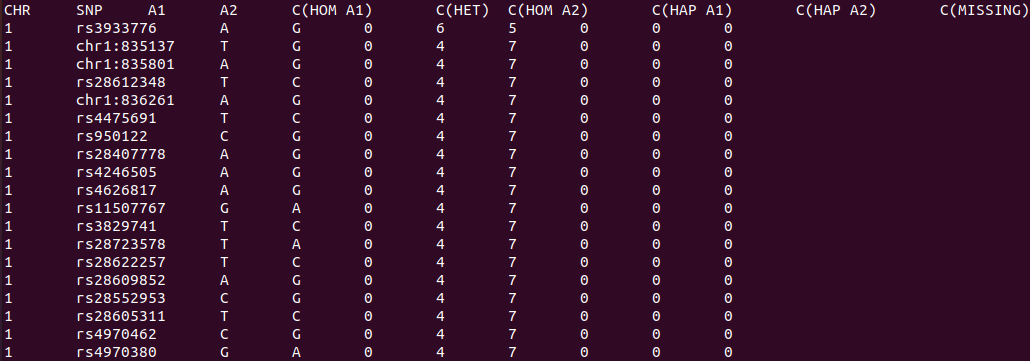
plink --bfile test_set --freqx --out freqxoutput : freqx.frqx
내용 : A1 , A2에 대해서 Homo, Hetero빈도수 확인이 가능합니다.
plink --bfile test_set --freq --out freqoutput : freq.frq
내용 : A1에 대한 AF (MAF)가 나오게 됩니다.

추가적으로 Case control을 지정을 하셧다면 (fam,map파일에서) case control에 대한 MAF확인도 가능합니다.
plink --bfile test_set --freq case-control --out freq1 --allow-no-sexoutput : freq1.frq.cc
내용 : case control 따로따로 MAF가 나옵니다. (Minor Allele는 1개 A1을 기준으로, A1이 plink에서는 minor allele입니다.)

2. --hardy
GWAS 주요 QC방법중 하나인 Hardy
plink --bfile test_set --hardy --out hardy --allow-no-sexoutput : hardy.hwe
내용 : Hardy-weinberg equilibrium에 대한 pvalue값을 줍니다.
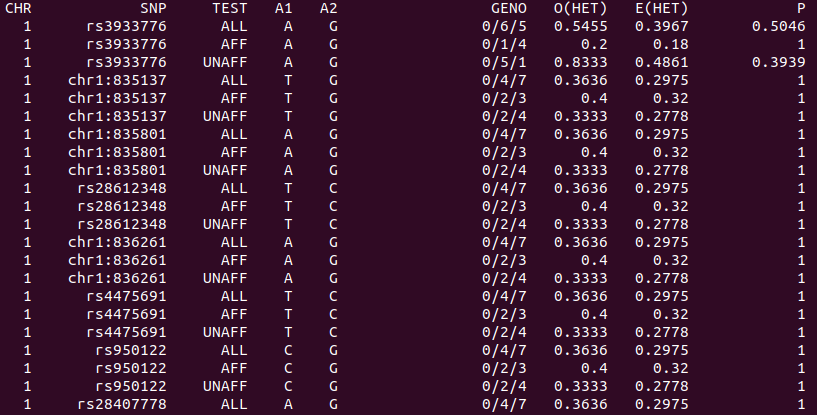
QC중에 Hardy로 cutoff 가 되지만, 그래도 잘되었는지 확인하기 위해서는 --hardy를 이용하시면됩니다.
오늘은 너무 늦게 글을 쓰느라, 내용도 조금 부실하게 준비를 했네요..
다음 포스팅도 plink에서 유용한 기능들에 대해 좀 더 알아보도록 하겠습니다.!!
'유전체 > GWAS' 카테고리의 다른 글
| [GWAS] GWAS Quality Control (QC) (4) | 2020.06.19 |
|---|---|
| [GWAS] GWAS 분석시 참고하기 좋은 사이트들 (0) | 2020.06.14 |
| [GWAS] plink 유용한 기능 (1) (exclude, extract) (1) | 2020.06.09 |
| [GWAS] plink 이용해서 vcf만들기~ (0) | 2020.06.08 |
| [GWAS] plink input format (형식확인) (0) | 2020.06.05 |


댓글