plink QC를 공부하기 전에 plink에서 유용한 기능들을 다루어 보도록하겠습니다.
QC는 그냥 default로 사용하는 -hwe 0.00001로 진행하고 --Geno 0.05 이런식으로는
알려드릴수 있으나, 그러면 공부하는 의미가 없죠! 그렇기 때문에 조금만 더 찾아 본 후에 포스팅 하도록 하겠습니다.
plink에 유용한 기능이 생각보다 많습니다.
초창기 plink 공부할 때, plink 사이트 들어가자마자, 원하는 기능들만 쏙 빼고 창을 닫았습니다. (물박사 종특ㅎㅎㅎㅎㅎㅎ)
왜냐하면, 영어로 된것들 다 읽기 귀찮았거든요... ㅎㅎㅎㅎㅎㅎㅎ
추후에 샘플들을 추가하거나, 혹은 분석한 것이 맞는지 확인을 할 때, 결국 다시 plink에 제가 필요한 기능들이 대부분 있더군요.
오늘 포스팅할 내용은 plilnk format을 합치는 기능!! 을 소개하고자 합니다.
GWAS가 population study라 그런지 샘플들의 수가 중요합니다.
그렇기 때문에, 샘플들 정보를 합치거나 나누는 것도 GWAS분석에서는 필수 기능들 중 하나일 것입니다.
plink에서는 plink format을 합치는 기능들이 있고, 원하는 변이들을 추출 혹은 제외하는 기능들이 있습니다.
1. --extract [뽑을 list파일]

plink --bfileplink --bfile ../ARHI_IMPUTED_CLEANED --extract Extract --out Extract --make-bedOutput : Extract bim, bed, fam

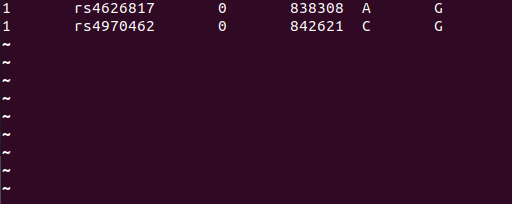
2. --exclude [제외시킬 list파일]
위 결과에서 exclude 할때 rs4626817 요놈만 지워보도록하겠습니다.
plink --bfile ./Extract --exclude exclude --out Exclude --make-bed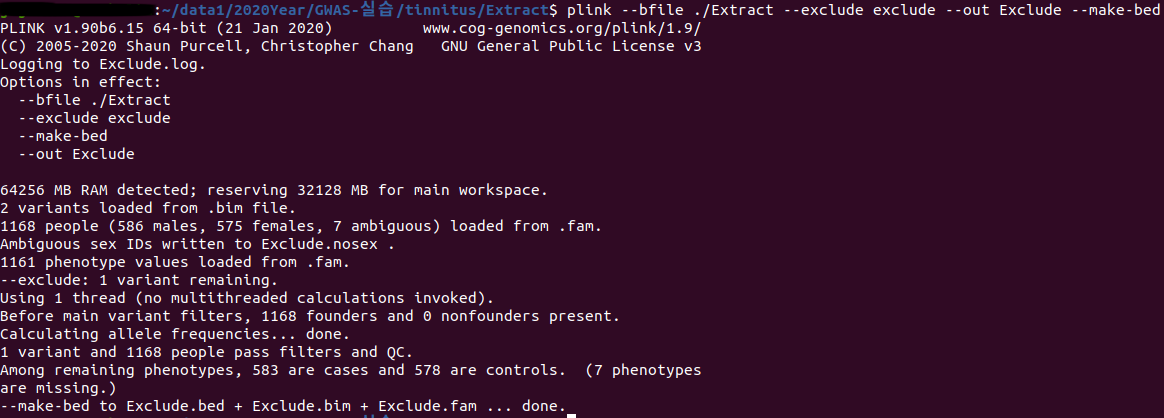

exclude를 진행하면 다음과 같이 제거가 된 채로 나오게 됩니다.
3. vcf to plink format & --merge, --bmerge
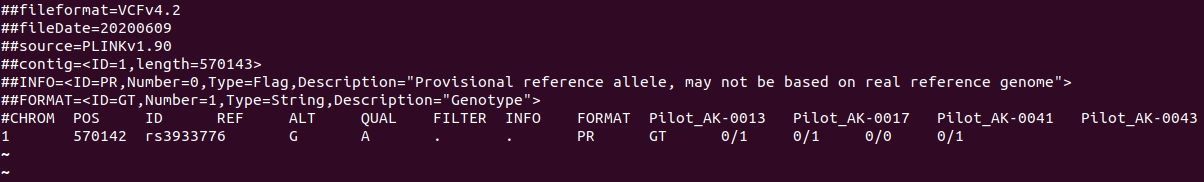
다음 두 개의 vcf가 있습니다. 같은 위치이고 변이도 같습니다.


위의 vcf 를 plink로 formatting하면,
plink --vcf test1.vcf --recode --out test1
plink --vcf test.vcf --recode --out test
그러면 map,ped파일들이 나오게 됩니다.
여기서 확인해야될 것은 map파일을 먼저 확인합니다.

이렇게 map파일 두번째 column이 동일할 때, 합칠 수가 있는데
plink --file test --merge test1.ped test1.map --out test_merge

이렇게 되면 binary output으로 합쳐진 plink파일을 확인할 수가 있습니다.
참고로! 순서 지켜주셔야 합니다.
--merge ped file, map file or --merge prefix text (test.ped,test.map 일 경우 test만 기재)
--bmerge bed file, bim file, fam file or --bmerge prefix text (test1.bed, test1.bim, test1.fam일 경우 test1만 기재)
주의 사항!
plink 의 default가 population내의 Minor allele frequency를 기준으로 잡기때문에
두 개의 Population을 합칠 때, 특히나 binary파일로 합칠때, bim파일이 서로 안맞는 경우가 있으니,
(minor(A1), major(A2)가 population 크기마다 다르게 나타날 수 있음.)
주의를 하시고, vcf -> plink format으로 변경할때, --keep-allele-order 추가하면 ref alt가 동일하게 formatting이 됩니다.
그리고 나서 merge로 합쳐주면 되죠. 그리고 간혹가다 map 2번째 column이 없는 경우! (bim에서도 2번째 column이 없는 경우 포함)
python이나 기타 기술로 채워주셔야지요~ 저는 rs number 아니면, chrom:pos:ref:alt 이런식으로해서 key를 만들어 넣습니다.
각자의 취향으로 진행하시면 될 것 같습니다.
오늘은 간단한 merge방법을 해보았습니다.
다음은 plink에서 할수있는 기본적인 통계! 를 배워보도록하겠습니다.~
감사합니다.
'유전체 > GWAS' 카테고리의 다른 글
| [GWAS] GWAS 분석시 참고하기 좋은 사이트들 (0) | 2020.06.14 |
|---|---|
| [GWAS] Plink 유용한 기능 (2) (freq, hardy) (3) | 2020.06.10 |
| [GWAS] plink 이용해서 vcf만들기~ (0) | 2020.06.08 |
| [GWAS] plink input format (형식확인) (0) | 2020.06.05 |
| [GWAS] plink 설치 및 설정하기. (0) | 2020.06.04 |



댓글