가장 먼저 fastq에서 alignment를 시켜줍니다. (bowtie2, bwa 등 이용해서 알아서)
그리고 sam file을 bam file로 변경시킴 (반대로 bam파일을 sam파일로 변경가능)
# sam to bam
samtools view -Sb [input.sam] > [output.bam]
# bam to sam
samtools view input.bam > output.sam
bam file에서 mapped read만 가져오기 위해서는 sam flags라는 것을 알아야 합니다.
https://broadinstitute.github.io/picard/explain-flags.html
Explain SAM Flags
broadinstitute.github.io
위 사이트를 들어가시게 되면 다음 고르는 곳이 있습니다. 그중에서 read unmapped를 이용합니다. (체크를 하면 옆에 summary에 값이 나옵니다. 우리가 사용해야할 값은 SAM Flag 값입니다.)

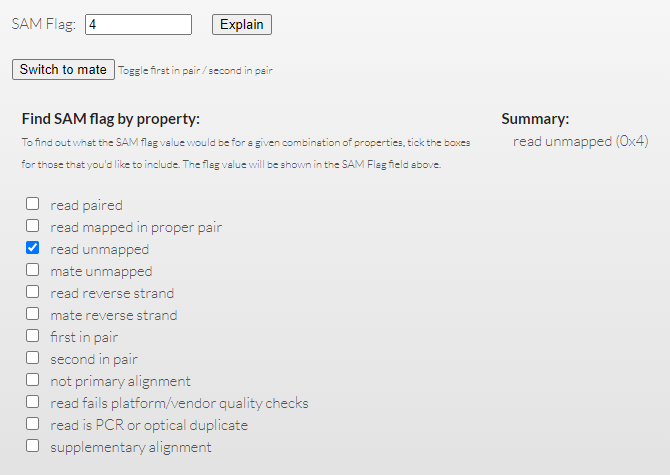
여기에서 SAM Flag에서 unmapped를 클릭해서 확인하면 SAM Flag에 4가 나오는데 이를 이용하여 samtools view를 한번 더 이용합니다.
# unmapped read 만 골라내기
samtools view -b -f 4 input.bam > output_unmapped.bam
# mapped read 만 골라내기
samtools view -b -F 4 input.bam > output_unmapped.bamsamtools view에 -b는 output이 bam file임을 알리는 것이고 -f는 SAM Flag에서 원하는 값을 가져오는 것이고, -F의 경우 SAM Flag 값을 제외한 나머지를 가지고 오게하는 Option입니다.
확인은 samtools idxstats를 이용해서 확인할 수가 있습니다. (추후 올리겠습니다.)
요즘 이것저것 분석을 하면서 오류를 덜 발생시키는 팁 하나를 간단하게 이야기하겠습니다.
(내용이 너무 짧아서 하나의 포스팅으로 하기에는 아닌것 같아서 짧게 설명하겠습니다.)
위에서도 output 나올때 결과파일을 samtools view -Sb input.sam > output.bam 이런식으로 '>' 를 이용합니다.
'>'의 경우 손쉽게 output을 저장 할수있는 방법입니다. (리눅스에서도 쉽게 파일을 만들수가 있죠 ex) ls > list 하면 list파일이 생기면서 안에 디렉토리 안에 있는 파일 이름들이 들어가 있습니다.)
그런데 이런 손쉬운 방법이 가끔 output을 변경할 수가 있습니다. 예를 들어 bowtie2, bwa의 경우 tools을 돌릴 때, 돌아가고 있는 표시가 나옵니다.
어디서 부터 align이 되고 있다 등의 정보들이요. 이런 정보들이 output에 섞여 들어갈수가 있습니다. 그렇기 때문에 tool을 잘 보시고 output option이 있는 경우 최대한 output option을 이용하시는 것을 추천드립니다. (이번에 저런일이 있어서 원인을 찾는데 조금 시간이 걸렸었습니다.)
'유전체' 카테고리의 다른 글
| [seurat] single cell 분석 시에 umap이 다르게 나오는 경우. (0) | 2022.05.23 |
|---|---|
| [R] ssgsea 의 scoring방법을 실습을 통해 이해해 보기 (0) | 2022.04.11 |
| [유전체] samtools, bedtools 이용해서 특정 부분 mapping된 fastq만들기 (0) | 2021.10.26 |
| [유전체] samtools 오류 해결! SEQ and QUAL are of different length (0) | 2021.08.24 |
| [유전체]samtools sort 에러 발생시 해결 (0) | 2021.08.17 |


댓글