1. Introduction
최근 제가 주목하고 있고, 실제 주식으로 보유하고 있는 다양한 회사들 중에서 제 나름대로 가장 기대가 큰 기업인 Quantum-SI에 대해서 이야기해보고자 합니다.
https://mopipe.tistory.com/170
[QSI] Quantum SI(QSI)에 투자하는 이유 (with. Jonathan M Rothberg)
1. Quantum si의 플랫폼은 중요하다. 단백질 sequencing의 경우 유전체분석에가 가장 중요합니다. 단백질은 실제 phenotype에 연관을 줄 정도로 중요합니다. 그러나 과학자들이 연구를 할때에는 대부분 mR
mopipe.tistory.com
이전에 포스팅했던 부분에서 QSI의 가장 중요한 기술들이 Recognizer, Aminopeptideases라고 이야기 했습니다. 저는 QSI에서 공개해놓은 이 Recognizer의 종류를 보고 20개 이상의 아미노산을 모두 커버할 수 없는데 이거 연구하는데 시간 오래걸리겠다 라는 생각을 했습니다.
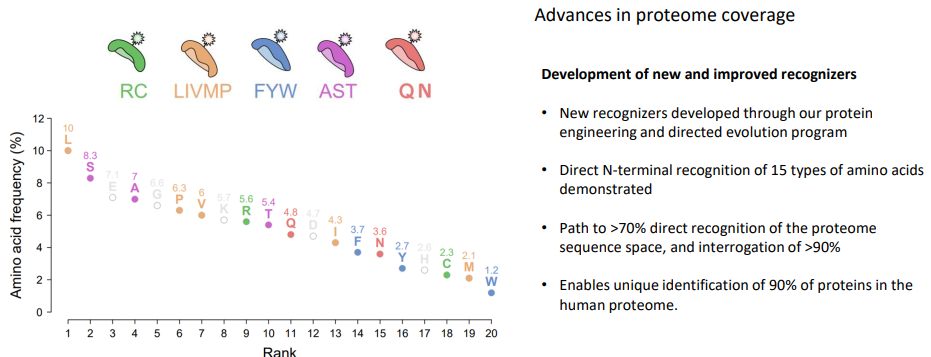
그리고 이 부분만 해결이 되면 정말로 상용화가 될 것 같다 라는 생각을 했었습니다. 그런데 이 자료를 본 이후 반년도 안되어서 단백질 시퀀싱을 상용화를 하겠다는 뉴스를 보게되어 깜짝 놀랬습니다. 한편으로는 기분이 좋기도 했고 다른 한편으로 걱정이 되기도 했죠. 왜냐하면 주가도 많이 떨어지기도 했고, 이 때문에 압박감으로 회사에서 결과물을 내놓는것 같았습니다. 아무튼... 상용화가 되었다길래 한번 내용좀 살펴봐야지 생각만 하다가 이제서야 보게 되었습니다.
2. 다시 한번 주입하는 단백질 시퀀싱의 중요성.
제가 QSI는 Recognizer가 20개의 amino acid 중에 15개라는 어마어마한 amino acid를 확인 할 수 있다고 이야기했습니다. 이것만으로도 정말 중요한 기술이라고 생각이 되었던 것이 최근 핫했던 면역항암제라고 들어보셨을까요?
면역항암제는 쉽게 이야기해서 우리 몸 내부에 있는 면역기능들을 활성화시켜서 암들을 치료시키려는 방법인데, 이때 면역항암제를 만드는데 다양한 루트가 있는데, 그중에서 Neoantigen과 연관된 neoepitope가 있습니다. 이 neoepitope은 특정 암에게서 공통적으로 발현이 될거라 생각되는 되는 하나의 단백질 조각인데 환자 내부에서 이 조각을 인식하는 T-cell들을 뽑아서 키워서 다시 환자에 주입하면 항암제가 됩니다. (자세한건 나중에 다시 포스팅... 할껍니다.. 아마... 돈이 되야 뭘 허지..)
뭐 암 백신도 비슷한 원리입니다. 저 조각들을 넣어서 하면 백신이 될 것이고, 암환자들의 경우는 암이 걸린 이유가 조각들을 인식하는 T-cell의 개수가 적어서 그런것 일 수 있으니 빼서 키워서 다시 주입시키는거구요(뭐 Common한 epitope이 없는 경우라면 Personalized epitope을 찾아서 질병을 고쳐야죠.).

여튼 말이 길어졌는데 여기서 저 조각 (neoepitope)을 확인하는 방법은 현재 두가지 정도입니다. DNA (RNA), 단백질로 확인이 가능한데, 암에서 특이 한 DNA에서 확인되었다 하더라도 실제 발현이 안되는 경우도 있을 것이고, RNA에서 발현이 되었어도 중요한건 단백질로 확인이 가능해야 합니다. 그렇기 때문에 주로 이러한 분석을 할때 사용하는 도구가 Mass Spectrometry (MS)입니다. 그런데 이 문제점이 neoepitope은 양이 적고 크기가 작은 경우가 다수여서 MS를 이용하더라도 감지가 안될 수 있다는 문제가 있습니다.
그런데 이 QSI 의 protein sequencing 방법을 이용하면 이 부분이 좀 더 쉽게 가능해 질 것으로 생각합니다. 아래에는 QSI ppt에서 보여주고 있는 그림입니다. 상용화가 되고 널리 사용하게 되면 다양한 연구가 이루어질 것이라고 생각됩니다.

3. Recognizer의 제한성.. 어떻게 해결할끈데??
최근 QSI 사이트를 이리 저리 둘러보다가 protein squencing에 대해 자세히 써놓은 pdf가 있는것을 발견했습니다. 아마 원래부터 있었던 것 같은데, 개인정보를 줘야지 받을 수 있는 부분이라 예전에 시도를 안해봤던 것 같습니다.
여기에서는 이야기 하는 것이 cloud based algorithms으로 결과를 낸다 라고 이야기하고 있습니다.
확실하게 알 수 있는 아미노산(Recognition segments,RSs)은 Pulse duration(PD)으로 나타낼 수 있습니다. 그리고 기기를 돌리게 되면 이 RS부분과 비인식부분(Non-recognition segment, NRS)에 대한 PD값들을 확인할 수가 있는데 QSI는 이러한 정보를 이용해서 유사한 peptide와 clustering 및 예측을 하게 됩니다(자체 PD database기반으로 분석할수있는 tool이 제공되는 듯합니다.). 이 예측시에는 QSI가 제작한 N terminal에서 시작하는 모든 가능한 4개의 amino acid에 대한 PD를 정확하게 예측할 수 있는 kinetic model을 같이 활용한다고 이야기하고 있습니다.
(We developed a kinetic model that accurately predicts the PD for every possible 4-amino-acid sequence that starts with an N-terminal recognizer target. 을 해석한 부분입니다.)
후보 단백질들도 여러가지 제시를 보여주는 것으로 나왔습니다.
4. 마무리
일단 QSI에서 protein sequencing이 상용화가 되어서 너무 좋고, 일단 상용화 후에 보편화까지 가봐야 장단점을 알 수 있을듯 합니다. 아쉬운점은 예측된 단백체라는점이 아쉽네요.. 그런데 MS도 마찬가지인 것으로 알고 있습니다. 그래도 방식을 보면 MS에 비하면 좀 더 혁신적이라도 생각이 드네요. 아마 1~2년정도의 시간이후에 사람들의 반응을 좀 봐야 될 것 같고, 논문 인용도 계속 tracking 해보면서 추후에 포스팅을 해보도록 하겠습니다.
감사합니다. 후원 문의는 주변에 아무 광고나 클릭이나 해줍쇼.
'경제공부 > 주식분석' 카테고리의 다른 글
| [Quantum-si, QSI] 단백실 시퀀싱업체 2024년 첫 발표자료 리뷰 (2) | 2024.04.11 |
|---|---|
| [QSI] Quantum SI(QSI)에 투자하는 이유 (with. Jonathan M Rothberg) (0) | 2022.03.31 |
| [주식] SoFi, QSi에 대한 생각 (0) | 2021.12.13 |
| 주식 현황 및 앞으로 계획 (0) | 2021.07.19 |
| 영화 엘리시움의 시대는 시작되었다. (0) | 2021.06.09 |


댓글